西工大新闻网7月6日电(卢慧甍 张轶峰 李迁)近日,生明学院卢慧甍副教授课题组研发首款昆虫嗅觉受体结构与功能预测的数据库(iORbase),相关研究成果以“iORbase: a database for the prediction of the structures and functions of insect olfactory receptors”为题发表在Insect Science期刊,该期刊是中科院1区TOP期刊,并隶属于“中国科技期刊卓越行动计划”的领军期刊。
昆虫是地球上多样性最丰富、分布最广的动物,在生态、农业、经济和健康等方面发挥着重要作用。昆虫嗅觉受体(Insect Olfactory Receptor,iOR)是昆虫聚集迁飞、寄主定位、警戒协助等行为及其强大的环境适应性与复杂的种群社会性的分子基础。由于iOR基因是多外显子编码且存在大量假基因,传统基因注释手段存在假阳性问题;此外,因为iOR属膜蛋白质且该家族成员数量庞大,开展功能实验难度大,iOR与小分子的识别机制相对匮乏。
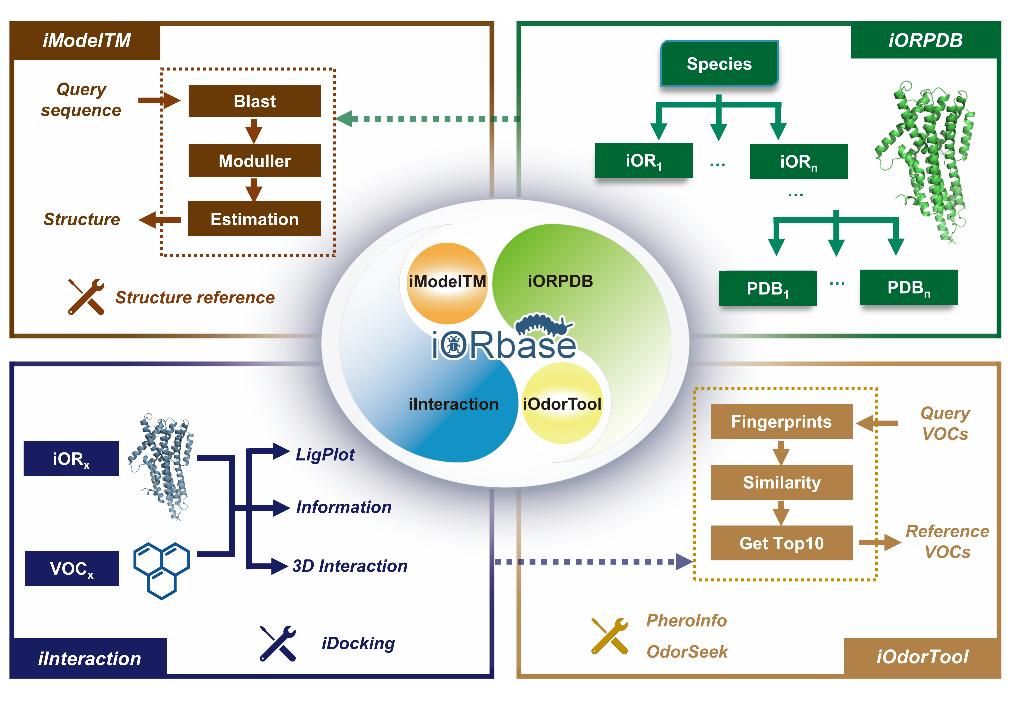
本论文将计算结构生物学方法应用于iOR的注释和结构功能预测,在传统基因注释基础上通过结构验证,重新注释了59个物种近6 000条iOR基因,预测了iOR的蛋白质空间结构,并计算了与2 077个信息素分子的对接数据,构建了“iOR结构与功能预测数据库——iORbase”。该数据库设计4个模块:蛋白质结构检索模块(iORPDB)、信息素对接查询模块(iInteraction)、iOR结构预测模块(iModelTM)和小分子功能预测模块(iOdorTool)。模块之间相互关联,其中iORPDB与iInteraction模块存储了iORbase数据库的核心数据,并分别为iModelTM和iOdorTool模块提供重要的数据支持,后者拥有更多应用工具,以满足用户多种使用需求。
iORbase数据库能够辅助昆虫学家在iOR功能实验中避免非功能蛋白的干扰,更理性地选择突变位点,更有效地解释昆虫生理实验的分子机制,从而为探索昆虫嗅觉编码机制,开展杀虫剂分子设计和仿生电子鼻研究等工作提供新的策略。
西北工业大学研究生李迁和张轶峰为共同第一作者,西北工业大学生命学院卢慧甍副教授、陕西师范大学生命科学学院李钢教授和徐畅副教授为论文通讯作者。本研究得到了国家自然科学基金、陕西省自然科学基金、中国空间站科学研究项目等资助。
(审核:杨慧)